TRANSCRIPTÓMICA
 Es el conjunto de ARN mensajero resultante de la
traducción del genoma bajo determinadas condiciones. Hay que tener cuenta que
en algunas células la actividad transcripcional es constante durante toda su
vida, pero en otras depende de estados fisiológicos o patológicos y estímulos
específicos.
Es el conjunto de ARN mensajero resultante de la
traducción del genoma bajo determinadas condiciones. Hay que tener cuenta que
en algunas células la actividad transcripcional es constante durante toda su
vida, pero en otras depende de estados fisiológicos o patológicos y estímulos
específicos.
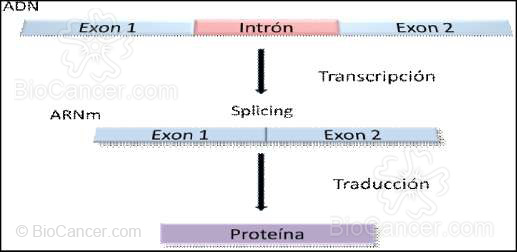
El splicing es
el mecanismo post-transcripcional en el que se cortan los intrones y se acoplan
los exones que codificarán las proteínas. Las mutaciones en las zonas de
empalme, heredadas o adquiridas, suelen ser el motivo del splicing aberrante
o patológico que produce un ARNm anómalo
Este fenómeno se ha
observado en genes implicados en carcinogénesis, como TP53, BRCA1, MLH1, FHIT o
TSG101 (Melver et al, 2000). Por ello, el estudio del splicing y
la caracterización de las variantes de éste adquieren importancia a la hora de
entender las transformaciones neoplásicas.
Microarrays o chips son
de gran utilidad para analizar simultáneamente un gran número de transcritos en
un solo ensayo, comparar muestras de tejido sano y afecto, y extraer
información del estado de la enfermedad a este nivel. Los patrones que
caracterizan cada estado podrán ser aplicados a la práctica clínica.
La transcriptomica es el estudio de los perfiles de expresión de todos los genes presentes en el genoma. El método
más utilizado es el de microarrays de DNA, que permite el análisis simultaneo
de la expresión de miles de genes.
Los microarrays de DNA
surgen de la necesidad de analizar la
cantidad de información procedente de
los grandes proyectos de secuenciación
de genomas.
Permiten elaborar mapas finos de transcripción
y proporcionan información indirecta de
los niveles de proteínas.
El análisis de microarrays de
DNA es una nueva tecnología que permite
estudiar simultáneamente la expresión de
miles de genes y analizar su expresión bajo
distintas condiciones experimentales. Los microarrays de DNA constan de miles
de conjuntos ordenados de moléculas de
DNA de secuencia conocida depositados en
un soporte sólido (~ 2 cm2) como
cristal, nylon o silicio. Cada
combinación (gen/muestra) se localiza de forma inequívoca en un punto del
microarray.
Como la genómica, la transcriptómica se vale de la bioinformática y las micromatrices. La idea básica de las micromatrices (o microarreglos) es
construir, sobre una membrana o lámina de vidrio, arreglos de muestras que
contienen fragmentos de ADN. Por otro lado se marca el ARN o el ADN copia
(cDNA) de una población celular con fluorescencia o radioactividad, y se usa
esta preparación para hibridar con el ADN de la micromatriz. Generalmente se
hibrida simultáneamente la misma micromatriz con una muestra de ARN o ADN copia
de referencia, para facilitar la comparación.
VIDEO SOBRE LA TRANSCRIPCIÓN DEL ADN QUE ESTA RELACIONADO CON LA TRANSCRIPTÓMICA